重度抑郁障碍(heavy depressive disorder,MDD)是全球疾病和残疾负担的主要因素,影响约5%的成人,全球疾病负担也在增加[1]。抗抑郁药治疗的疗效有限。此外,由于缺乏MDD的生物标志物,需要依赖临床访谈,导致误诊率很高[2]。尽管具有重大的社会和经济影响,但MDD的分子机制仍未得到充分理解。有人认为,表观遗传机制可能在暴露于不良生活事件后介导抑郁症的风险。表观遗传学包括通过转录、转录后、翻译或翻译后过程控制的基因表达的稳定改变,包括DNA修饰、染色质重塑、组蛋白修饰、RNA修饰和非编码RNA(ncRNA)调控,而DNA序列没有任何变化。表观遗传机制介导暴露于负面生活经历后对压力的易感性的持久升高,并为遗传和环境因素的整合提供了机制框架[3]。
近期,华中科技大学同济医学院陈建国教授课题组在Current Opinion In Neurobiology(CONB)上发表了题为“Epigenetic mechanisms in depression: Implications for pathogenesis and treatment”的综述文章。研究人员探讨了抑郁症表观遗传学领域的最新研究进展,评估了表观遗传变化作为抑郁症诊断和治疗生物标志物的潜力。发现不良生活事件会改变不同脑细胞的表观遗传修饰,包括神经元、小胶质细胞和星形胶质细胞。表观遗传修饰与抑郁症相关的表观遗传特征的重塑有关。细胞特异性表观遗传调控揭示了抑郁症发病机制的特定因果关系。目前关于抑郁症表观遗传生物标志物的证据,需特别关注DNA甲基化和ncRNA表达。

一、三个脑区的全长转录组概述
表观遗传学是指基因表达的广泛遗传变化,这些变化是由于环境影响而发生的,而不是由于DNA序列的改变。表观遗传机制介导暴露于负面生活经历后对压力的易感性的持久升高,并为遗传和环境因素的整合提供了机制框架。这篇综述系统地总结了近年来在理解表观遗传调控在抑郁症中的作用和潜在机制方面的进展(图1)。
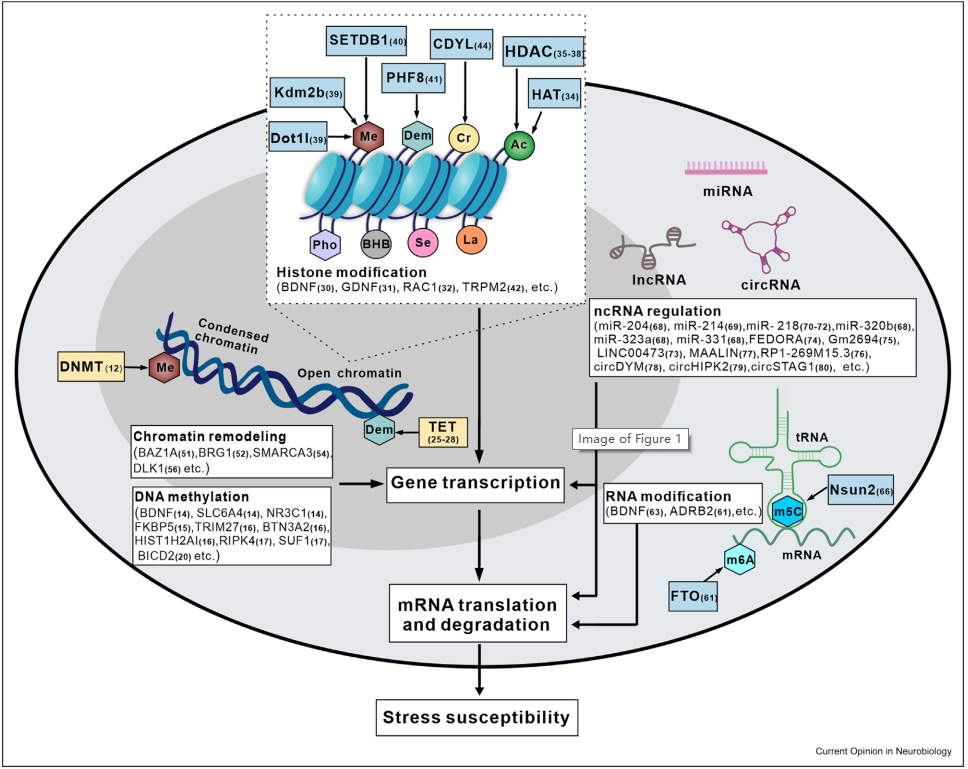
图1与抑郁症相关的主要表观遗传变化。(图源:Chen HS,et al.,Curr Opin Neurobiol,2024)
二、DNA甲基化调节抑郁症的进展
DNA甲基化是由称为DNA甲基转移酶(DNMT)的酶催化的。迄今为止,已经确定了五种不同的DNMT。通常,启动子中CpG岛的甲基化增强会抑制基因表达,而甲基化减少会促进基因表达。DNMTs控制DNA甲基化模式的建立和保存,DNMTs的活性调节情绪行为。DNA甲基化组范围关联研究(MWAS)显示,一些CpG甲基化位点涉及抗原加工和免疫反应,如TRIM27、BTN3A2和HIST1H2AI,它们与应激反应性有关[4]。母体抑郁与RIPK4基因和SUF8基因启动子(chr70378380:70378994-8和chr70378380:70378995-1)的DNA甲基化水平升高有关[5]。非CpG甲基化在成熟神经元中显著表达,其上调与突触发育和突触密度增加相吻合[6]。此外,早期逆境通过影响非CpG甲基化和组蛋白修饰之间的相互作用,破坏杏仁核内的免疫系统过程和小GTP酶信号通路[7]。然而,非CpG甲基化对抑郁症病理生理学的贡献需要进一步研究。DNA去甲基化涉及两种不同的机制:被动去甲基化和主动去甲基化。当5mC通过复制稀释丢失时,就会发生被动去甲基化,这是由于抑制DNA甲基化维持和5mC水平降低所致。DNA甲基化的位置影响抑郁症的病理过程(表1)。
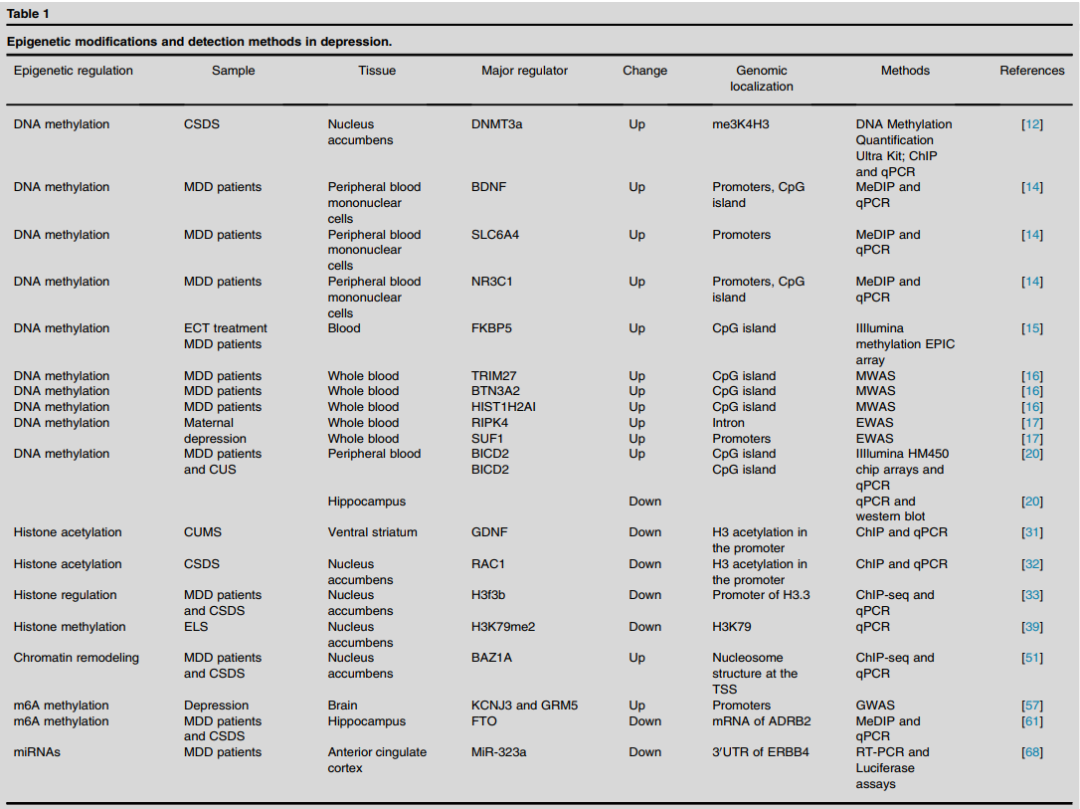
表1.抑郁症的表观遗传修饰和检测方法(表源:Chen HS,et al.,Curr Opin Neurobiol,, 2024)
三、组蛋白修饰在抑郁症发病机制中的应用
在真核细胞中,核小体构成染色质的基本重复结构单元,其中核心组蛋白(H2A、H2B、H3和H4)和组蛋白相关蛋白与DNA一起包装。组蛋白修饰通过影响核小体稳定性、染色质介导的过程和组蛋白之间的相互作用,在抑制或激活基因表达中发挥作用。几种组蛋白修饰通过多种机制与抑郁症的发病机制有关。组蛋白乙酰化(HAc)涉及通过添加乙酰基团对组蛋白H3和H4上的赖氨酸(K)残基进行共价修饰。抑郁症的病理生理学与组蛋白H3泛乙酰化的整体减少有关。复制非依赖性组蛋白变异体,包括H3.3变异体,在调节活动依赖性神经元转录中起着重要作用,并与抑郁症有关。NAc中H3.3增加的动力学对于调节基因表达以响应异常的社会压力至关重要,导致小鼠抑郁样行为的发展[8]。HAc受组蛋白乙酰转移酶(HAT)调节,可促进DNA-组蛋白相互作用的松动并暴露基因启动子区域。相比之下,组蛋白去乙酰化酶(HDACs)可增强这些相互作用,保护基因启动子区域并抑制基因转录和表达[9]。在探索组蛋白修饰时,考虑不同大脑区域的变异和神经元特异性至关重要。例如,丙咪嗪治疗导致NAc中HDAC5的表达增加,而海马中HDAC5的过表达阻碍了丙咪嗪的抗抑郁功效。这种差异可能是因为NAc主要由GABA能中棘神经元(MSN)组成,而海马体主要由锥体神经元组成。
四、染色质重塑与抑郁症有关
在结构紧密的异染色质中,翻译过程受到阻碍,而结构松散的常染色质促进翻译。组蛋白的修饰会改变染色质结构,从而影响基因的转录可及性。此外,ATP依赖性染色质重塑复合物通过ATP水解动态调节染色质可及性,影响基因转录活性。利用ATP的染色质组装和重塑因子(ACF)是ISWI家族中研究最广泛的蛋白质复合物。在MDD患者和CSDS模型小鼠中,ISWI亚基BAZ1A及其组装的ACF复合物在NAc中的表达升高。ACF复合物通过调节转录起始位点(TSS)的核小体结构来抑制基因表达,从而增加应激易感性。然而,对ATP依赖性染色质重塑复合物的研究仍处于早期阶段,许多未解决的问题仍然存在。需要进一步研究抑郁症中染色质重塑复合物的亚基组织、组装、三维结构和相互作用伙伴的综合表征。
五、RNA修饰参与抑郁症的发展
RNA修饰,也称为表观转录组修饰,已被发现在转录组水平上广泛存在。在各种RNA修饰中,N6-甲基腺苷(m6A)是最普遍的mRNA修饰,它调节mRNA的成熟、降解、输出和翻译效率,涉及广泛的生物过程。m6A的调节功能主要由三个同源因子执行:甲基转移酶(写入者)、去甲基化酶(橡皮擦)和效应蛋白(读取者),一些文献表明,改变的m6A修饰在突触功能障碍和抑制中起着重要作用。在人脑中观察到的m6A谱显示,具有脑特异性m6A修饰的基因在突触和神经元通路中功能富集,这与抑郁症有关。此外,m6A对环境刺激有反应,表明其在桥接分子和行为对压力的反应中起着重要作用。FTO基因(一种重要的RNA去甲基化酶)的水平受行为压力的影响。据报道,FTO通过降低Adrb2 mRNA中的m2A水平来增加肾上腺素受体β6(ADRB2)的表达,随后促进突触发生并减轻雄性小鼠的抑郁样行为。FTO也被确定为三环类抗抑郁药(TCA)的靶标。
表观转录组修饰在大脑中转移(t)RNA中的作用引起了越来越多的兴趣。tRNA是70-90个核苷酸的小RNA,受到43种不同类型的表观转录组学修饰的高度调控。最近的研究揭示了以前被低估的tRNA m5C修饰对神经元功能和情绪的影响[10]。M6A和m5C不仅存在于mRNA和tRNA中,还存在于rRNA和ncRNA中。因此,慢性应激引发的RNA修饰在转录和翻译水平上都会显着影响基因表达。m6A mRNA修饰的变化可能有助于抑郁症的治疗反应。
六、ncRNA调控参与抑郁症的过程
ncRNA,包括microRNA(miRNA)、长链非编码RNA(lncRNA)和环状RNA(circRNA),在调节抑郁症的发病机制方面至关重要。miRNA不仅能够与3'UTR结合,还能够与5'UTR、编码序列和启动子结合,从而对基因表达产生抑制作用。一些研究调查了抑郁症个体或动物模型的脑组织样本中的候选miRNA,揭示了参与各种途径的miRNA,如多巴胺系统、BDNF信号传导和单胺能神经传递。抑郁症患者中30%的差异表达基因是lncRNA。例如,LINC00473,一种在抑郁症中具有性别特异性作用的lncRNA,位于神经元核中,在抑郁症女性的皮层中下调。LncRNAs也与抑郁性自杀的危险因素有关。在死于抑郁自杀的个体的喙前扣带回皮层(rACC)中对lncRNA表达进行转录组范围的分析,揭示了lncRNA的显着失调。
由于通过转录组分析鉴定了人类和动物抑郁症模型中异常的circRNA表达,circRNAs在抑郁症发病机制中的参与引起了相当大的关注。Yuan等人最近发现,circRNA DYM(circDYM)在MDD患者的外周血和抑郁症动物模型的海马体中均下调。circDYM表达降低导致miR-9释放,随后下调HECTD1,导致HSP90泛素化抑制和小胶质细胞活化增加[11]。此外,海马星形胶质细胞中circRNA的存在与抑郁症的进展有关。CUS暴露后,海马体中circRNA HIPK2(circHIPK2)的水平显着升高。敲除星形胶质细胞中的circHIPK2通过减轻星形胶质细胞功能障碍来减轻CUS诱导的小鼠抑郁样行为[12]。许多研究已经确定了ncRNA表达的性别差异和细胞特异性(见表2)。此外,相同的RNA可以在不同的大脑区域表现出不同的作用。因此,在研究ncRNA时,应仔细关注性别差异、脑区和细胞特异性。
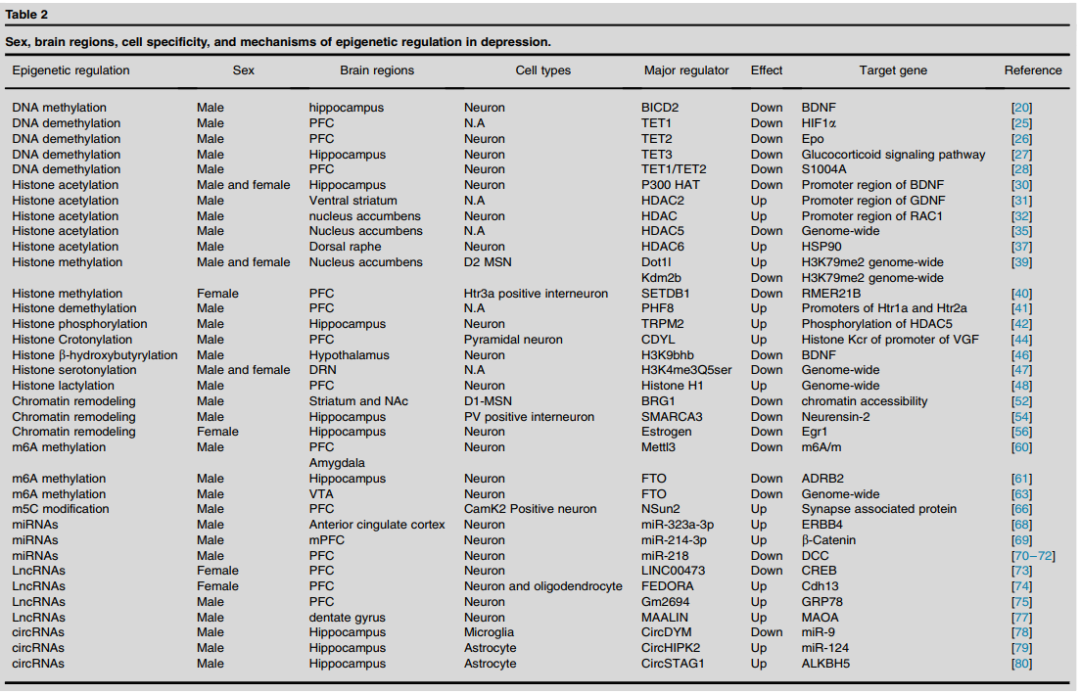
表2.性别、大脑区域、细胞特异性和抑郁症表观遗传调控机制(表源:Chen HS,et al.,Curr Opin Neurobiol,, 2024)
七、表观遗传学在抑郁症中的临床应用
表观遗传生物标志物在抑郁症的早期诊断中起着关键作用(见表3)。DNA甲基化的生物标志物已经过广泛的研究。外周血中BICD2的甲基化与MDD的风险呈正相关,而BDNF启动子I的甲基化与抑郁风险降低相关。ncRNA也可以作为抑郁症的表观遗传生物标志物。有几种miRNA被确定为MDD的潜在生物标志物,包括miR-204-5p、miR-320b、miR-323a-3p和miR-331-3p,这些生物标志物在MDD患者的死后脑组织中被发现改变。在表观遗传调控的研究中,正在进行的发现突出了某些表观遗传变化作为诊断和治疗抑郁症靶点的潜力。特别是血液中DNA甲基化和ncRNA的改变显示出作为诊断抑郁症和预测抑郁症治疗反应的分子标志物的巨大前景。
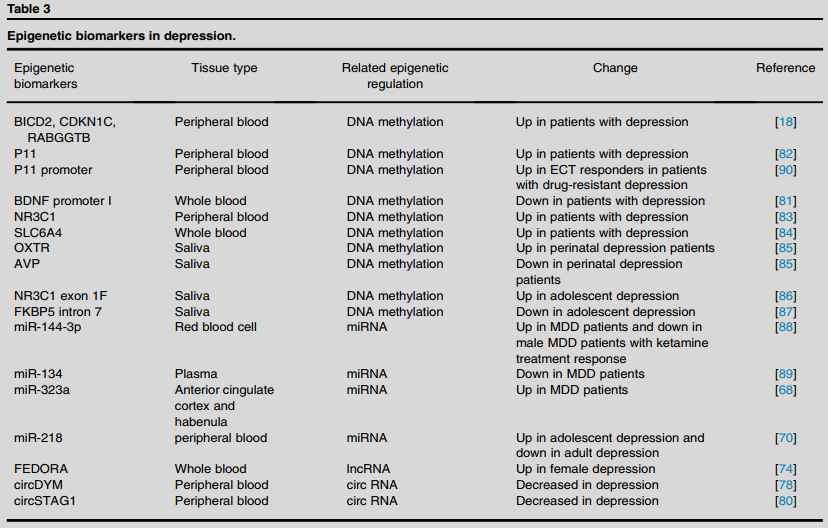
表3.抑郁症的表观遗传生物标志物(表源:Chen HS,et al.,Curr Opin Neurobiol, 2024)
八、总结与展望
MDD的风险受遗传和环境因素的影响。基因与环境的相互作用是由表观遗传机制决定的,可能是抑郁症的主要致病因素。这篇综述首先阐明了过去5年中涉及DNA甲基化、组蛋白修饰、RNA修饰和ncRNA的机制,以及它们在抑郁症中的作用,部分涉及细胞类型特异性表观遗传调节。此外,根据临床应用总结了抑郁症的诊断和治疗相关生物标志物。全面掌握抑郁症的表观遗传机制对于产生诊断和治疗抑郁症的创新概念和策略至关重要。由于抑郁症中表观遗传调控的复杂性,迫切需要先进的分析方法和模型,例如单细胞测序和机器学习。此外,必须通过全基因组研究来加强对与抑郁症相关的表观遗传机制的理解,特别关注整个生命周期的性别和细胞类型特异性。总之,通过全基因组研究,特别是在整个生命周期中特别强调性别和细胞类型特异性,促进对抑郁症表观遗传机制的理解,将促进MDD诊断和治疗干预措施的发展。
原文链接:https://doi.org/10.1016/j.conb.2024.102854
参考文献1.Disease GBD, Injury I, Prevalence C: Global, regional, and national incidence, prevalence, and years lived with disability for 354 diseases and injuries for 195 countries and territories, 1990–2017: a systematic analysis for the Global Burden of Disease Study 2017. Lancet 2018, 392:1789–1858.2.Mitchell AJ, Vaze A, Rao S: Clinical diagnosis of depression in primary care: a meta-analysis. Lancet 2009, 374:609–619.3.Klengel T, Binder EB: Epigenetics of stress-related psychiatric disorders and gene x environment interactions. Neuron 2015, 86:1343–1357.4.Shen X, Caramaschi D, Adams MJ, Walker RM, Min JL, Kwong A, Hemani G, Genetics of DNAMC, Barbu MC, Whalley HC, et al.:DNA methylome-wide association study of genetic risk for depression implicates antigen processing and immune responses. Genome Med 2022, 14:36.5.Drzymalla E, Crider KS, Wang A, Marta G, Khoury MJ, Rasooly D: Epigenome-wide association studies of prenatal maternal mental health and infant epigenetic profiles: a systematic review. Transl Psychiatry 2023, 13:377.6.. Lister R, Mukamel EA, Nery JR, Urich M, Puddifoot CA, Johnson ND, Lucero J, Huang Y, Dwork AJ, Schultz MD, et al.: Global epigenomic reconfiguration during mammalian brain development. Science 2013, 341:1237905.7.Lutz PE, Chay MA, Pacis A, Chen GG, Aouabed Z, Maffioletti E, Theroux JF, Grenier JC, Yang J, Aguirre M, et al.: Non-CG methylation and multiple histone profiles associate child abuse with immune and small GTPase dysregulation. Nat Commun 2021, 12:1132.8.Lepack AE, Bagot RC, Pena CJ, Loh YE, Farrelly LA, Lu Y, Powell SK, Lorsch ZS, Issler O, Cates HM, et al.: Aberrant H3.3 dynamics in NAc promote vulnerability to depressive-like behavior. Proc Natl Acad Sci U S A 2016, 113:12562–12567.9.Wu MS, Li XJ, Liu CY, Xu Q, Huang JQ, Gu S, Chen JX: Effects of histone modification in major depressive disorder. Curr Neuropharmacol 2022, 20:1261–1277.10.Blaze J, Navickas A, Phillips HL, Heissel S, Plaza-Jennings A, Miglani S, Asgharian H, Foo M, Katanski CD, Watkins CP, et al.: Neuronal Nsun2 deficiency produces tRNA epitranscriptomic alterations and proteomic shifts impacting synaptic signaling and behavior. Nat Commun 2021, 12:4913.11.Zhang Y, Du L, Bai Y, Han B, He C, Gong L, Huang R, Shen L, Chao J, Liu P, et al.: CircDYM ameliorates depressive-likebehavior by targeting miR-9 to regulate microglial activation via HSP90 ubiquitination. Mol Psychiatry 2020, 25:1175–1190.12.Zhang Y, Huang R, Cheng M, Wang L, Chao J, Li J, Zheng P, Xie P, Zhang Z, Yao H: Gut microbiota from NLRP3-deficient mice ameliorates depressive-like behaviors by regulating astrocyte dysfunction via circHIPK2. Microbiome 2019, 7:116